Carla Columbano de Oliveira – Laboratório de Controle Pós-Transcricional de Expressão Gênica
O principal objetivo do nosso laboratório é compreender as vias de controle pós-transcricional de expressão gênica em eucariotos, focando principalmente em dois processos principais, maturação ribossomal e splicing de mRNA. Visamos caracterizar a função molecular de fatores proteicos envolvidos nessas vias de processamento, usando como modelo a levedura Saccharomyces cerevisiae. Dada a conservação evolutiva destes processos em eucariotos, os resultados poderão ser depois extrapolados para humanos, nos quais doenças já foram identificadas como sendo causadas por mutações nos genes das proteínas ortólogas estudadas por nós. Temos vagas no laboratório para alunos de mestrado e doutorado e para pós-doutorandos. Interessados em se candidatar a uma vaga, podem acessar o nosso site para mais detalhes e escrever para o e-mail abaixo.
https://www.carlaoliveiralab.com/
Daniela Ramos Truzzi - Laboratório de Mecanismos Redox do Óxido Nítrico
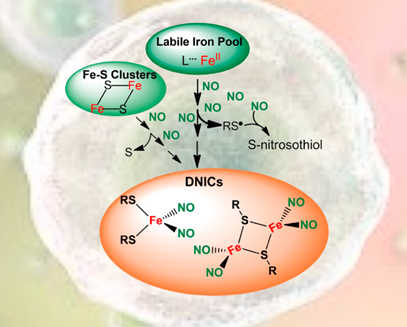
Em nosso Laboratório estudamos os metabólitos do óxido nítrico (NO). O NO é uma molécula sinalizadora envolvida em uma série de processos fisiopatológicos. O conhecimento de como o NO e seus metabólitos atuam tem permitido o desenvolvimento de diversas intervenções farmacológicas ao longo dos anos. Recentemente, foi demonstrado que, entre todos os metabólitos de NO, os dinitrosilos complexos de ferro (DNICs) são os mais abundantes no meio intracelular. Dessa forma, nosso Laboratório busca compreender os mecanismos que levam à formação de DNICs em células, identificar as macromoléculas que os constituem e, por fim, avaliar suas possíveis funções biológicas. Os resultados obtidos contribuirão para elucidar como o NO é armazenado em células e fundamentar aplicações farmacológicas de DNICs no futuro. Temos vagas para alunos de mestrado e doutorado. Para maiores informações, envie um email para dtruzzi@iq.usp.br
https://publons.com/researcher/2565837/daniela-r-truzzi/publications/
https://doi.org/10.1021/acs.inorgchem.1c00823
Deborah Schechtman - Laboratório de Bioquímica da Sinalização Celular
O principal objetivo do laboratório de bioquímica da sinalização celular é compreender as vias de sinalização envolvidas na dor para o desenvolvimento de analgésicos não-opióides. Visamos estudar as vias de sinalização envolvidas na embriogênese das vias da dor, e na transmissão de sinais da dor inflamatória pelo fator de crescimento neural NGF ativando a quinase TrkA, e o papel da PKMz nos processos de remodelamento das sinapses na dor crônica. Temos vagas no laboratório para alunos de iniciação científica, mestrado, doutorado e pós-doutorado. Caso tenham interesse em se candidatar a uma vaga visite o nosso site e marque uma conversa (deborah@iq.usp.br)
http://www.iq.usp.br/portaliqusp/?q=pt-br/users/d%C3%A9borah-schechtman
Evandro Araújo de Souza - Laboratório de Homeostase Proteica
Estamos em busca de alunos de pós-graduação para desenvolver o projeto intitulado "O que promove o rejuvenescimento celular na linhagem germinativa, e isso pode ser replicado em células somáticas?". O estudo utilizará o nematoide Caenorhabditis elegans como modelo experimental e abordará questões relacionadas ao envelhecimento e à agregação proteica, investigando vias da rede de proteostase, como a resposta à proteínas mal-enoveladas no Retículo Endoplasmático (Unfolded Protein Response, UPR). O projeto empregará diversas técnicas, incluindo RNAi, microscopia de fluorescência e CRISPRa.
Para mais informações, acesse http://sites.usp.br/evandro ou entre em contato por e-mail: evandro@iq.usp.br.
Fabio Luis Forti - Laboratório de Sinalização de Sistemas Biomoleculares (LSSB)
As respostas de células eucarióticas frente a agentes endógenos e exógenos que causam danos no DNA (genotóxicos) dependem do acionamento rápido e eficiente de vias de sinalização e/ou redes integradas de sinalização que levam ao reconhecimento e reparo do material genético lesado possibilitando a manutenção metabólica, sobrevivência e proliferação celular. Transdução de Sinais, Regulação do Ciclo Celular e Mecanismos Moleculares de Manutenção da Estabilidade Genômica das células são os principais interesses de nosso laboratório! Abordagens Bioquímicas, Proteômicas, de Biologia Celular e Molecular, e de Biologia de Sistemas focadas em redes de Interação Proteína-Proteína (Interactomas) são usadas para se investigar enzimas tirosina quinases, tirosina fosfatases duais e gtpases que controlam, por mecanismos ainda pouco conhecidos, as respostas de linhagens celulares normais e tumorais humanas (leucemia, melanoma, glioma, câncer de cervix, mama, pulmão e próstata) a agentes externos (físicos e químicos) causadores de danos no DNA genômico. Um maior entendimento e conhecimento de eixos, vias ou redes de sinalização intracelular por estas enzimas (e outras) nos levarão ao melhor planejamento e intervenção molecular que possam viabilizar tratamentos terapêuticos usando pré-sensibilização celular combinada de quimioterápicos, associada ou não a radioterapia, para contra-atacar os diferentes tipos de câncer humanos de maior incidência e/ou mortalidade na população.
O Laboratório de Sinalização de Sistemas Biomoleculares (LSSB) oferece oportunidades a estudantes de graduação vindos de diferentes áreas de formação (químicos, biólogos, biomédicos, farmacêuticos, matemáticos, físicos, médicos e outros) relacionadas aos interesses científicos do laboratório para realizarem sua pós-graduação (Mestrado / Doutorado). Candidatos motivados, pró-ativos, que gostam de enfrentar desafios, aprender conceitos teóricos e práticos são bem-vindos! Experiência prévia laboratorial é desejável, mas não totalmente necessária. Os interessados devem enviar e-mail diretamente para o docente Professor Fábio Forti (flforti@iq.usp.br), apresentando seus interesses, além de CV e uma carta de recomendação.
Glaucia Mendes Souza - Laboratório de Proteômica Aplicada à Processos Inflamatórios
Venha fazer parte do Projeto Temático FAPESP “Genômica e Biotecnologia para o Aumento da Produtividade, Resiliência a Climas Futuros e Bioprodutos de Cana”!
Sobre o Projeto:
Nosso projeto foca na cana-de-açúcar e cana-energia integrando sustentabilidade com biotecnologia. O objetivo é desvendar os mecanismos moleculares que promovem a alta biomassa e a tolerância à seca, além de explorar novas vias metabólicas para a produção de bioprodutos. Venha trabalhar em um ambiente dinâmico e colaborativo, onde ciência e inovação caminham lado a lado!
Sobre o Laboratório de Bioenergia e Sustentabilidade:
O Laboratório de Bioenergia e Sustentabilidade, sob a coordenação da Profa. Glaucia Souza no IQ-USP, é um centro pioneiro em pesquisa e inovação, que usando genômica, foca em abordagens avançadas e sustentáveis para a produção de biomassa, biocombustíveis e bioprodutos. Com infraestrutura moderna e equipe multidisciplinar, o laboratório é referência em genômica, genômica funcional, biotecnologia, bioinformática e uso sustentável dos recursos avaliando também o impacto climático e o potencial de mercado de novas rotas tecnológicas.
Bolsas FAPESP já Disponíveis:
- Doutorado Direto - Bioprodutos: Conduza pesquisas sobre rotas metabólicas e extração supercrítica de compostos de interesse. O projeto será em colaboração com o Prof. Pio Colepicolo do IQ-USP. Os requisitos estão detalhados em https://fapesp.br/bolsas/dd.
- Doutorado Direto - Biomassa: Enfrente o desafio da seca com análises fisiológicas, morfológicas e RNA-Seq, construindo redes de coexpressão de genes de cana. O projeto será em colaboração com a Profa. Monalisa Carneiro da UFSCar de Araras. Os requisitos estão detalhados em https://fapesp.br/bolsas/dd.
Se você é movido pela paixão pela pesquisa e deseja fazer parte dessa revolução sustentável, não perca essa chance! Candidate-se agora e venha transformar o futuro da bioenergia!
Os candidatos deverão enviar para o e-mail clembke@iq.usp.br: currículo ou link para currículo Lattes, histórico escolar de graduação e certificado de conclusão da graduação e indicação para qual vaga está aplicando.
Nicolas Carlos Hoch – Laboratório de Estabilidade Genômica
Regina Lucia Baldini - Laboratório da Regulação da Expressão Gênica em Microrganismos
Sistemas de Sinalização em Bactéria
Nosso grupo tem como modelo de estudo a bactéria Pseudomonas aeruginosa, um patógeno oportunista com alta resistência a antibióticos. Os projetos em andamento incluem avançar no entendimento de vias de sinalização pelo segundo mensageiro c-di-GMP, que está envolvido com a transição das células móveis para células sésseis, que se organizam em forma de biofilmes. Entretanto, a multiplicidade de proteínas envolvidas na síntese e degradação deste nucleotídeo em P. aeruginosa implica em funções mais específicas destas proteínas, que almejamos dissecar usando estratégias genéticas e bioquímicas. Outros projetos incluem a resposta a condições ambientais que aumentam a adaptação da bactéria à presença de antibióticos no meio. Temos vagas para estudantes de pós-graduação e para pós-doutorado. Para maiores informações, visite http://iq.usp.br/portaliqusp/?q=pt-br/users/regina-l%C3%BAcia-baldini ou mande uma mensagem para baldini@iq.usp.br
Ricardo José Giordano - Laboratório de Bioquímica Combinatorial
O laboratório de Bioquímica Combinatorial tem 2 vagas de doutorado direto com bolsas FAPESP. Nosso grupo estuda a formação de vasos sanguíneos na saúde e na doença (p.ex., câncer e retinopatias) em busca de novos medicamentos para as chamadas doenças dependentes da angiogênese. Para uma das bolsas, conhecimento básicos de computação e programação são recomendados para utilizar ferramentas computacionais para varredura de compostos (fármacos). Para mais informações, visitar a página do laboratório (https://giordanolab.com/ ou h
Roberto Kopke Salinas – Laboratório de RMN de Biomoléculas
Os projetos de pesquisa desenvolvidos pelo grupo envolvem uma abordagem multidisciplinar que combina bioquímica e biologia molecular, espectroscopia, e computação, para estudar estrutura, dinâmica e interações de sistemas biológicos. Através destes estudos pretende-se compreender do ponto de vista atômico e molecular como proteínas desempenham a sua função biológica. Atualmente estamos buscando alunos interessados em estudar a estrutura e a função de proteínas de membrana e regiões intrinsecamente desordenadas. São de particular interesse os trocadores de Na+/Ca2+ (NCX) eucariótico e procariótico, o trocador de Na+/Ca2+ mitocondrial (NCLX), e o sistema de secreção de macromoléculas bacteriano T4SS. Alunos interessados em fazer mestrado ou doutorado podem obter mais informações sobre as linhas de pesquisa do grupo no link ou no email abaixo.
Solange Maria de Toledo Serrano – Instituto Butantan
No Laboratório de Toxinologia Aplicada do Instituto Butantan, a Dra. Solange Maria de Toledo Serrano estuda a bioquímica de peptídeos e proteínas presentes nos venenos de diversas serpentes. Tem caracterizado estruturalmente e funcionalmente as enzimas proteolíticas presentes nesses venenos e empregado estratégias proteômicas para auxiliar a desvendar importantes aspectos da evolução de toxinas animais. Atualmente, o grupo se dedica a elucidar os efeitos locais e sistêmicos do envenenamento por Bothrops jararaca. As técnicas utilizadas são essencialmente imunofluorescência, Western blot e espectrometria de massas orientada para peptidômica e proteômica.
https://orcid.org/0000-0001-8994-1904
https://www.cetics.com.br/en/pesquisadores/solange-serrano/
https://butantan.gov.br/pesquisa/ddc/venenos-envenenamentos-e-toxinas-de-venenos